Može li NIPT pogriješiti? – detaljne informacije
https://journals.plos.org/plosone/article/false-negative-NIPT-results
Neinvazivno prenatalno testiranje (NIPT) pokazalo je male šanse za lažno negativan rezultat. Budući da “fetalna” DNK u krvi majke potječe iz citotrofoblasta korionskih resica (CV), neki lažno negativni rezultati imat će biološko podrijetlo.
Na temelju svojeg iskustva s citogenetskim studijama CV-a, centar Cees Oudejans, (VU University Medical Center u Nizozemskoj pokušao je procijeniti ovaj rizik. 5967 CV uzoraka trudnoća s visokim rizikom za uobičajene aneuplodije citogenetski je ispitano u centru između siječnja 2000. i prosinca 2011. Svi slučajevi fetalne trisomije 13, 18 i 21 retrospektivno su proučavani na prisutnost normalnog kariotipa ili mozaicizma < 30% u kratkotrajno uzgojene (STC-) resice. Od 5967 uzoraka pronađena su 404 slučaja trisomije 13, 18 i 21 (6,8%). Od ova 404 slučaja, 14 (3,7%) imalo je normalan ili nizak mozaični kariotip u STC-resicama i stoga bi potencijalno bili propušteni s NIPT-om. Uključivalo je 2% (5/242) svih slučajeva trisomije 21 i 7,3% (9/123) svih slučajeva trisomije 18. U 1:426 (14/5967) NIPT će potencijalno propustiti trisomije 18 ili 21 kod pacijentica s visokim rizikom za uobičajene aneuploidije zbog biološkog fenomena odsutnosti kromosomske aberacije u citotrofoblastu.
Validacija neinvazivnog prenatalnog testiranja (NIPT) za otkrivanje fetalne trisomije otkrila je da postoji mala vjerojatnost lažno pozitivnog i lažno negativnog rezultata [1b]. Iako tehnička ograničenja mogu objasniti ove lažne rezultate, lažni rezultati mogu imati i biološku osnovu. Činjenica da je „fetalna“ DNK ustvari izvanstanična DNK u frakciji plazme majke koja potječe iz citotrofoblasta korionskih resica (CV) objašnjava barem dio odstupanja između rezultata NIPT-a i stvarnog fetalnog kariotipa [2-4].
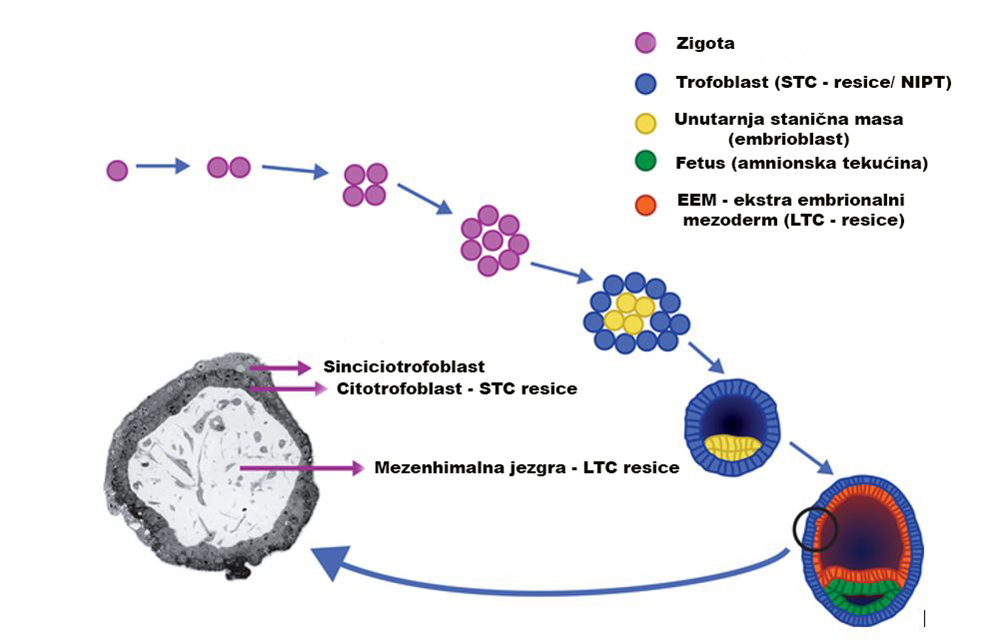
Slika 1. Trofoblast oblikuje resice koje prodiru u maternicu i razvijaju se u posteljicu; stanice trofoblasta se razvijaju u citotrofoblast, koji omogućuje implantaciju te konačno i razvoj posteljice i sinciciotrofoblast koji stvara korijalni gonadotropin od 10. Dana. Resice i posteljica stvaraju trofičke hormone te pružaju arterijsku i vensku izmjenu između cirkulacija zametka i majke.
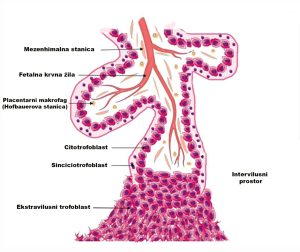
Kako bismo razumjeli biološko podrijetlo lažno pozitivnih i lažno negativnih rezultata NIPT-a, važno je razumjeti citogenetiku korionskih resica. CV se može obraditi za citogenetičke studije na dva načina: izravnom ili poluizravnom tehnikom koja se naziva kratkotrajno uzgojene resice (STC-resice) i metodom dugotrajne pripreme koja se naziva dugotrajno uzgojene resice (LTC-resice) [7]. Podrijetlo stanica koje se istražuju u citogenetskim preparatima bitno je različito u objema tehnikama: stanice u STC-resicama potječu iz citotrofoblasta, vanjskog sloja stanica CV-a, a one u LTC-resicama su pretežno iz unutarnjeg sloja stanica. Zlatni standard za citogenetsku analizu CV-a je istraživanje STC (citotrofoblast)- i LTC (mezenhimalna jezgra) resica [8]. S NIPT-om se istražuje samo DNA iz citotrofoblasta i stoga će rezultati biti usporedivi s onima iz STC-resica.
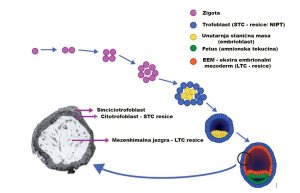
Slika 2. Rani embrionalni razvoj od zigote do blastociste.
Citotrofoblast koji se proučava u kratkotrajno uzgojenim resicama (STC-resice) i s NIPT-om potječe iz trofoblasta blastociste, dok mezenhimalna jezgra, istražena u dugotrajno uzgojenim resicama (LTC-resice) potječe iz ekstra- embrionalnog mezoderma blastociste (EEM). I EEM i fetus potječu od unutarnje stanične mase (ICM) blastociste.
https://doi.org/10.1371/journal.pone.0146794.g001
Može li NIPT pogriješiti? – detaljne informacije
Već osamdesetih godina kromosomske analize STC- i LTC-resica otkrile su da se kromosomska konstitucija citotrofoblasta može razlikovati od one mezenhimske jezgre i fetusa [9]. To je posljedica različitog embrionalnog porijekla triju spomenutih dijelova. Citotrofoblast potječe od trofoblasta blastociste, dok mezenhimalna jezgra CV-a kao i sam fetus potječu od unutarnje stanične mase (ICM) (Slika 2). Točnije, mezenhimalna jezgra potječe iz hipoblasta ICM-a, a sam fetus iz epiblasta [10, 11]. Pogreške postzigotne mitotske diobe u kromosomski normalnim kao i abnormalnim embrijima dovest će do kromosomskog mozaicizma koji se nalazi u oko 1 do 2% CV [12]. Zbog neravnomjerne raspodjele abnormalnih stanica u različitim odjeljcima, kariotipovi citotrofoblasta, mezenhimalne jezgre i fetusa mogu biti različiti [13]. U većini ovih slučajeva mozaika kromosomski abnormalna stanična linija ograničena je na citotrofoblast i/ili mezenhimalnu jezgru CV-a, dok sam fetus ima normalan kariotip [8, 12]. Taj se fenomen naziva ograničeni placentni mozaicizam (CPM) [14] i čini 72% svih slučajeva mozaicizma otkrivenog CV-em [15]. Od tri moguća tipa CPM-a, onaj s (mozaičnom) kromosomskom aberacijom ograničenom na citotrofoblast i nije prisutan u mezenhimalnoj jezgri ili fetusu, naime CPM tip 1, je najrašireniji. CPM tip 1 uzrokovat će lažno pozitivan NIPT rezultat ako je postotak abnormalnih stanica u citotrofoblastu dovoljno visok [16]. Isto vrijedi i za manje prevalentni CPM tip 3 s kromosomski abnormalnim stanicama ograničenim na oba stanična sloja CV-a i odsutnim u samom fetusu.
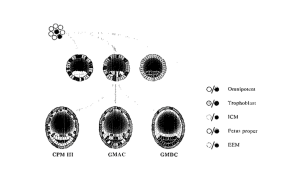
Slika 3. Primjer moguće distribucije normalnih i abnormalnih stanica u slučaju mozaicizma koji dovodi do teoretskih kombinacija prema Pittalisu ct al. (1994)
Carolina Dina Francina van den Berg
Quality and Reliability of Prenatal Cytogenetics
S druge strane, kromosomski mozaicizam također može dovesti do lažno negativnih rezultata NIPT-a. Dvije vrste mozaicizma povezane su s normalnim kariotipom u citotrofoblastu, dok sam fetus ima kromosomsku aberaciju. Prvi, generalizirani mozaicizam ograničene izravne normalnosti generalised mosaicism confined direct normality (GMDD)[12], karakteriziran prisutnošću kromosomske aberacije u fetusu i mezenhimalnoj jezgri posteljice, pri čemu je citotrofoblast kromosomski normalan. Drugi, iako iznimno rijedak, ograničeni fetalni mozaicizam (CFM) s normalnim kariotipom u STC- i LTC-resicama i s krajnjim abnormalnim citogenetskim rezultatima u fetusu [12, 17]. Obje vrste mozaicizma pokazat će normalne rezultate NIPT-a zbog normalnog kariotipa u citotrofoblastu, iako fetus ima abnormalni kariotip. Tipovi generaliziranog mozaicizma s apsolutnom ili relativnom podudarnošću (GMAC ili GMRC, respektivno), potencijalno također mogu proći neotkriveni s NIPT-om, ako je postotak abnormalnih stanica u STC-resicama ispod određenog praga [18] (Slika 3. i 4.).
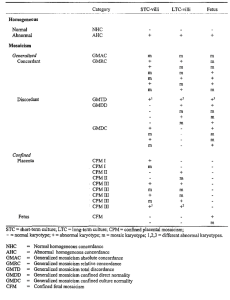
Slika 4. Klasifikacija teoretskih kombinacija mozaicizama prema Kalouscku i sur. (1992) i
Pittalis i sur. (1994)
U kliničkoj praksi sada je općenito prihvaćeno da su, ako NIPT otkrije trisomiju 13, 18 ili 21, potrebna dodatna ispitivanja invazivnim postupkom, ponajprije amniocentezom, kako bi se potvrdili rezultati, jer NIPT nije u potpunosti dijagnostički [19-21]. ]. To će otkriti lažno pozitivne rezultate i time se može spriječiti nepotreban prekid trudnoće. Međutim, jednako je važno shvatiti da ako je rezultat NIPT normalan, postoji mogućnost da je taj rezultat lažno negativan.
Može li NIPT pogriješiti? – detaljne informacije
Malo je informacija dostupno u vezi s lažno negativnim rezultatima NIPT, osim nekoliko prijavljenih slučajeva [1]. Studije o lažno negativnim rezultatima NIPT-a zahtijevale bi temeljita praćenja svih testiranih trudnoća, uključujući citogenetička ispitivanja onih koje su rezultirale pobačajem ili intrauterinom smrću fetusa. Kako bismo proučili rizik od biološkog lažno negativnog rezultata NIPT-a koji uključuje kromosome 13, 18 i 21, u centru Cees Oudejans, (VU University Medical Center u Nizozemskoj) retrospektivno su istražen svi CV slučajevi fetalne trisomije 13, 18 i 21 tijekom 12-godišnjeg razdoblja (siječanj 2000.-prosinac 2011.). Svi slučajevi fetalne trisomije 13, 18 i 21 među 5967 CV uzoraka koji su citogenetski ispitani u centru proučavani su na prisutnost normalnog kariotipa ili niske razine mozaicizma < 30% u STC-resicama što bi potencijalno doveli do lažno negativnih rezultata NIPT-a. Prag od 30% odabran je na temelju prethodno objavljenih radova [16, 22]. Glavne indikacije za uzimanje uzoraka CV-a u ovim slučajevima trisomije bile su ultrazvučne abnormalnosti fetusa, povećana nuhalna translucencija, starija dob majke > 36 godina i/ili abnormalni rezultati probira u prvom tromjesečju (rizik >1:200).
Ova se studija poklapa sa studijom Van den Berga i sur. (2006.) koji opisuje rezultate u 2389 CV uzoraka rađenih u razdoblju od studenog 2000. do srpnja 2005.
Unatoč našem standardnom protokolu tijekom razdoblja istraživanja u kojem smo rutinski izvodili STC- kao i LTC-resice, u većini slučajeva trisomije 21 analizirane su samo STC resice jer se ova kromosomska aberacija može smatrati “određenom abnormalnošću” kada se nađe u STC-resicama , bez obzira na indikaciju[8]. Slično tome, u većini slučajeva potpuno razvijene trisomije 13 i 18, kariotipizacija je bila ograničena na onu STC-resica ako su fetalne ultrazvučne anomalije, otkrivene u vrijeme uzimanja CV uzoraka, odgovarale kromosomskoj aberaciji.[8] U svim ostalim slučajevima (normalni ili mozaični rezultati u STC-resicama ili 100% trisomija 13 ili 18 u STC-resicama bez ultrazvučnih abnormalnosti),također su ispitivane LTC-resice. GTG banding je rutinski korišten u svim slučajevima. Proučavano je najmanje 8 metafaza u STC-resicama (raspon 8-21) i 10 u LTC-resicama (raspon 10-25). Dodatno, u većini slučajeva mozaika proveden je FISH na interfaznim jezgrama kako bi se isključila prisutnost niske razine mozaicizma u kariotipski normalnim STC-resicama, ili kako bi se dalje proučavala razina mozaicizma u mozaičnim STC i/ili LTC-slučajevima.
Može li NIPT pogriješiti? – detaljne informacije – Rezultati
Na 5967 CV uzoraka nađeno je ukupno 404 (6,7%) fetalnih trisomija 13, 18 i 21. Svi ovi slučajevi smatrani su pravim slučajevima fetalne trisomije zbog:
- prisutnost 100% trisomije u LTC-resicama
- prisutnost 100% trisomije 13 ili 18 u STC resicama u vezi s fetalnim ultrazvučnim abnormalnostima koje odgovaraju abnormalnom kariotipu
- prisutnost 100% trisomije 21 u STC-resicama, bez obzira na indikaciju kako su opisali van den Berg et al. [8, 23].
Od ova 404 slučaja, 14 (3,7%) imalo je normalan (N = 9) ili nisko-mozaični (<30%) (N = 5) kariotip u STC-resicama i stoga bi potencijalno bili propušteni da je NIPT izveden u tim slučajevima (Stol 1). To znači da će u 14/5967 visokorizičnih trudnoća trisomija potencijalno biti izostavljena s NIPT-om na temelju bioloških razloga.
Rasprava
Izvanstanična ‘fetalna’ DNK u krvi majke potječe iz citotrofoblasta [2] i nije uvijek u skladu s pravom fetalnom DNK. Ovo će objasniti barem neke od lažno pozitivnih i lažno negativnih NIPT rezultata koji su trenutno objavljeni. Nakon uvođenja biopsije korionskih resica (CV) kao alternative amniocentezi ranih osamdesetih, kratko su se vrijeme u mnogim svjetskim laboratorijima citogenetička istraživanja korionskih resica izvodila isključivo na preparatima citotrofoblasta [5]. Međutim, ubrzo nakon njegovog uvođenja saznali smo da kariotip ovog sloja stanica, koji potječe iz trofoblasta blastociste [10], nije uvijek reprezentativan za kariotip fetusa. To je zbog kromosomskog mozaicizma s neravnomjernom distribucijom normalnih i abnormalnih stanica u različitim odjeljcima (trofoblast, ekstraembrionalni mezoderm i fetus) konceptusa [12, 24]. Ovaj fenomen doveo je do objavljivanja mnogih radova o lažno pozitivnim i negativnim rezultatima CV-a i do promjene protokola koji zagovara korištenje dviju metoda kulture: STC-resice koje istražuju citotrofoblast i LTC-resice koje istražuju mezenhimsku jezgru CV-a. Pokazalo se da će samo analiza obje metode dovesti do visokog stupnja točnosti [9]. Uvođenjem NIPT-a povijest se ponovila. Štoviše, zbog podrijetla ‘fetalne’ izvanstanične DNK i mogućnosti kromosomskog mozaicizma, 100% osjetljivost i specifičnost za NIPT možda se i nikada neće postići.

Tablica 1. Slučajevi trisomije 18 s normalnim ili niskim mozaikom (<30%) rezultiraju STC resicama u našoj kohorti od 5967 CV uzoraka.
https://doi.org/10.1371/journal.pone.0146794.t003
Znajući da s NIPT-om istražuje citotrofoblast CV-a, brojke rizika za lažno pozitivne i lažno negativne rezultate NIPT-a trebale bi se izvesti iz CV-brojki. Stoga je Centar prove retrospektivnu analizu svojih CV podataka 5967 pacijenata s visokim rizikom za uobičajene aneuploidije, uglavnom AMA > 36 godina, ftCT (abnormalni rezultati kombiniranog testa u prvom tromjesečju > 1:200), povećanu NT i druge ultrazvučne abnormalnosti. Prikupljeni su slučajevi kromosomskog mozaicizma s normalnim rezultatima ili niske razine mozaicizma < 30% u STC-resicama i potpuno razvijenom trisomijom 13, 18, 21 u LTC-resicama i/ili fetusu kako bi se odredila potencijalna učestalost lažno negativnih rezultata NIPT-a. Studija je pokazala da se u 0,2% (14/5967) (95% CI 0,13%-0,39%) pacijenti koji imaju visok rizik za uobičajene aneuploidije mogu očekivati lažno negativan rezultat NIPT za trisomiju 13, 18 i 21. Čini se da je ova brojka veća od brojki za lažno negativne STC resice navedene u literaturi. Ovo je djelomično objašnjeno uključivanjem slučajeva niskih mozaika u prezentirane podatke, tako da bi radi kvalitetne usporedbe ove slučajeve mozaika po mogućnosti trebalo isključiti. Učinivši to, lažno negativna stopa u našoj seriji zapravo bi i dalje bila 0,15% (9/5967) (95% CI 0,07%-0,29%) što je u skladu s našim prethodno objavljenim podacima (Van den Berg et al. 2006), ali bi i dalje bio viši od podataka u nekoliko drugih objavljenih izvještaja. Najvjerojatnije objašnjenje je razlika u indikacijama za uzimanje uzoraka koja se pokazuje manjom incidencijom trisomija 13, 18 i 21 u radu Grati i sur. [25] (3% u odnosu na 7% (404/5967) u našoj skupini pacijenata). To potvrđuje i rad Toutaina i sur. [26, 27] koji su pronašli incidenciju GMDD-a od 1/386 (0,26%) u malim serijama CV uzorkovanih za povećanu nuhalnu translucenciju ili druge ultrazvučne abnormalnosti. Dakle, ovisno o tome postoji li visok ili nizak rizik za aneuploidiju, procijenjena šansa za lažno negativnu dijagnozu NIPT varirat će između 0,02% i 0,26% (Tablica 1.). Ipak, treba uzeti u obzir nekoliko stvari. Prvo, ove brojke su vjerojatno precijenjene zbog dobro poznate varijacije placente u slučajevima mozaicizma. Stoga se ne može isključiti da CV biopsija u prvom tromjesečju može propustiti citogenetski abnormalne kotilidone koji će vjerojatno dovesti do abnormalnih rezultata NIPT-a pod pretpostavkom taloženja cijelog placentnog citotrofoblasta u majčinu cirkulaciju. Međutim, po našem mišljenju, rizik smatramo minimalnim jer su svi uzorci rađeni kontinuirano praćeni ultrazvučno te rađeni putem transabdominalne aspiracije umjesto transcervikalnom aspiracijom. Drugo, na temelju trenutnog iskustva [16, 22] očekuje se da će mozaik manji od 30% biti propušten s NIPT-om. Međutim, to će sigurno uvelike ovisiti o fetalnoj frakciji s potencijalno izostavljenom 100% trisomijom ako je fetalna frakcija preniska i mozaikom od ≤ 30% otkrivenim ako je fetalna frakcija visoka.
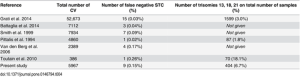
Tablica 2. Lažno negativni citogenetski rezultati u STC-resicama koje uključuju trisomiju 13, 18 i 21: pregled literature.
https://doi.org/10.1371/journal.pone.0146794.t004
Posebice je očigledan rizik za lažno negativnu trisomiju 18, s tim da je u ovoj studiji 7,3% svih fetalnih slučajeva s trisomijom 18 potencijalno propušteno ako je proveden NIPT. Preferencijalna uključenost trisomije 18 u GMDD također se može prepoznati u nedavnom radu Grati et al. [25] (6 od 382 slučaja (1,6%)), ali i u dosadašnjoj CV literaturi (ACC CVS baza podataka 2005). Zapravo, Porreco et al. [28] koji su proveli prospektivnu multicentričnu opservacijsku studiju uspoređujući NIPT s rezultatima invazivnog testiranja u 3430 slučajeva s visokim rizikom od uobičajenih aneuploidija, otkrili su da 3 od 38 slučajeva trisomije 18 (7,9%) ima lažno negativan rezultat NIPT-a.
Razumno je misliti da stvarna prevalencija lažno negativnih NIPT rezultata može biti čak i veća od spomenutih brojki. Lažno negativni rezultati zbog tehničkih problema kao što je niska fetalna frakcija, ili na primjer kao posljedica visokog BMI [29, 30, 31] ili zbog nekih fetalnih aneuploidija [32] dobro su poznati problem (dodatak: kod NIPT-ova koji koriste counting metodologiju) i treba ih dodati ovim brojkama. S druge strane, vjerojatno se može zanemariti rizik od lažno negativnog NIPT rezultata zbog ograničenog fetalnog mozaicizma (CFM). Ova vrsta mozaicizma iznimno je rijetka, vjerojatno manja od 0,003%, iako apsolutne brojke nisu dostupne zbog nepotpunih naknadnih istraživanja [17].
Do sada je dostupno malo informacija o stvarnoj prevalenciji lažno negativnih NIPT rezultata. Ipak se objavljuje sve veći broj izvješća o slučajevima na ovu temu [1]. Stoga, na temelju procijenjenog rizika od neotkrivanja 3,5% (14/404) fetalnih trisomija 13, 18 i 21 s NIPT-om, kao što je prikazano u ovom radu, preporučuje se naknadno citogenetičko testiranje u svim slučajevima s normalnim rezultatima NIPT-a:
- ako se ultrazvukom otkriju fetalne anomalije
- ako dođe do spontanog pobačaja ili intrauterine smrti te
- ako se rodi sindromski abnormalno dijete.
U takvim slučajevima želimo potaknuti citogenetičko testiranje fetusa ili djeteta i placente. To će u konačnici dovesti do boljeg znanja o stvarnoj prevalenciji lažno negativnih rezultata NIPT.
Zaključno, retrospektivna analiza ~6000 CV uzoraka pacijenata s visokim rizikom od aneuploidije otkrila je da u 1:426 (14/5967) trudnoća fetalna trisomija 13, 18, 21 potencijalno može biti propuštena s NIPT-om zbog odsutnosti kromosomske aberacije u citotrofoblastu. Štoviše, bez obzira na indikaciju, mogli bismo izračunati da 2% (5/242) odnosno 7,3% (9/123) trisomija 21 i 18 vjerojatno neće biti otkriveno NIPT-om zbog kromosomski normalnog ili niskog mozaičkog citotrofoblasta. Zbog relativno malog broja slučajeva, za trisomiju 13 ova brojka ostaje nejasna. Budući da će većina trisomičnih fetusa, osobito onih s trisomijom 13 ili 18, pokazati anomalije koje se mogu otkriti ultrazvukom, dio tih lažno negativnih rezultata otkrit će se kasnije tijekom trudnoće ili će završiti intrauterinom smrću. Važno je da pacijenti koji se odluče za NIPT budu obaviješteni o tehničkim i biološkim ograničenjima neinvazivnog prenatalnog postupka kako bi se mogao napraviti informirani izbor između neinvazivnog ciljanog testiranja i invazivnog uzorkovanja s citogenetskim testiranjem CV-a ili amniocita [33]. ]. U međuvremenu, temeljita naknadna istraživanja u slučajevima normalnih rezultata NIPT-a u konačnici će dovesti do pravih brojki za osjetljivost NIPT-a.
Tanja iz Panorame
1.a Bianchi DW, Parker RL, Wentworth J, Madankumar R, Saffer C, Das AF, et al.
DNA sequencing versus standard prenatal aneuploidy screening. N Engl J Med 2014;370:799–808.
1.b Bianchi DW, Wilkins-Haug L. Integration of noninvasive DNA testing for aneuploidy into prenatal care: what has happened since the rubber met the road? Clin Chem. 2014;60(1):78–87. Epub 2013/11/21. doi: clinchem.2013.202663 [pii] pmid:24255077.
Flori E, Doray B, Gautier E, Kohler M, Ernault P, Flori J, et al. Circulating cell-free fetal DNA in maternal serum appears to originate from cyto- and syncytio-trophoblastic cells. Case report. Hum Reprod. 2004;19(3):723–4. Epub 2004/03/05. [pii]. pmid:14998976.
- Srebniak MI, Diderich KE, Noomen P, Dijkman A, de Vries FA, van Opstal D. Abnormal non-invasive prenatal test results concordant with karyotype of cytotrophoblast but not reflecting abnormal fetal karyotype. Ultrasound Obstet Gynecol. 2014;44(1):109–11. Epub 2014/03/04. pmid:24585494.
- Faas BH, de Ligt J, Janssen I, Eggink AJ, Wijnberger LD, van Vugt JM, et al. Non-invasive prenatal diagnosis of fetal aneuploidies using massively parallel sequencing-by-ligation and evidence that cell-free fetal DNA in the maternal plasma originates from cytotrophoblastic cells. Expert Opin Biol Ther. 2012;12 Suppl 1:S19–26. Epub 2012/04/17. pmid:22500971.
- Simoni G, Brambati B, Danesino C, Rossella F, Terzoli GL, Ferrari M, et al. Efficient direct chromosome analyses and enzyme determinations from chorionic villi samples in the first trimester of pregnancy. Hum Genet. 1983;63(4):349–57. Epub 1983/01/01. pmid:6862440.
- Gibas LM, Grujic S, Barr MA, Jackson LG. A simple technique for obtaining high quality chromosome preparations from chorionic villus samples using FdU synchronization. Prenat Diagn. 1987;7(5):323–7. Epub 1987/06/01. pmid:2956585.
- Smidt-Jensen S, Christensen B, Lind AM. Chorionic villus culture for prenatal diagnosis of chromosome defects: reduction of the long-term cultivation time. Prenat Diagn. 1989;9(5):309–19. Epub 1989/05/01. pmid:2657708.
- van den Berg C, Van Opstal D, Brandenburg H, Wildschut HI, den Hollander NS, Pijpers L, et al. Accuracy of abnormal karyotypes after the analysis of both short- and long-term culture of chorionic villi. Prenat Diagn. 2000;20(12):956–69. Epub 2000/12/13. [pii]. pmid:11113907.
- Sachs ES, Jahoda MG, Los FJ, Pijpers L, Reuss A, Wladimiroff JW. Interpretation of chromosome mosaicism and discrepancies in chorionic villi studies. Am J Med Genet. 1990;37(2):268–71. Epub 1990/10/01. pmid:2248296.
- Crane JP, Cheung SW. An embryogenic model to explain cytogenetic inconsistencies observed in chorionic villus versus fetal tissue. Prenat Diagn. 1988;8(2):119–29. Epub 1988/02/01. pmid:3362778.
- Bianchi DW, Wilkins-Haug LE, Enders AC, Hay ED. Origin of extraembryonic mesoderm in experimental animals: relevance to chorionic mosaicism in humans. Am J Med Genet. 1993;46(5):542–50. Epub 1993/06/15. pmid:8322818.
- Pittalis MC, Dalpra L, Torricelli F, Rizzo N, Nocera G, Cariati E, et al. The predictive value of cytogenetic diagnosis after CVS based on 4860 cases with both direct and culture methods. Prenat Diagn. 1994;14(4):267–78. Epub 1994/04/01. pmid:8066036.
- Wolstenholme J. Confined placental mosaicism for trisomies 2, 3, 7, 8, 9, 16, and 22: their incidence, likely origins, and mechanisms for cell lineage compartmentalization. Prenat Diagn. 1996;16(6):511–24. Epub 1996/06/01. pmid:8809892.
- Kalousek DK, Barrett I. Confined placental mosaicism and stillbirth. Pediatr Pathol. 1994;14(1):151–9. Epub 1994/01/01. pmid:8159612.
- Battaglia P, Baroncini A, Mattarozzi A, Baccolini I, Capucci A, Spada F, et al. Cytogenetic follow-up of chromosomal mosaicism detected in first-trimester prenatal diagnosis. Prenat Diagn. 2014;34(8):739–47. Epub 2014/03/19. pmid:24633594.
- Gao Y, Stejskal D, Jiang F, Wang W. False-negative trisomy 18 non-invasive prenatal test result due to 48,XXX,+18 placental mosaicism. Ultrasound Obstet Gynecol. 2014;43(4):477–8. Epub 2013/11/05. pmid:24186002.
- Wolstenholme J, Evans J, English C. A 45,X fetus with false-negative 46,XX findings in CVS. Prenat Diagn. 2008;28(3):266–7. Epub 2008/02/28. pmid:18302306.
- Canick JA, Palomaki GE, Kloza EM, Lambert-Messerlian GM, Haddow JE. The impact of maternal plasma DNA fetal fraction on next generation sequencing tests for common fetal aneuploidies. Prenat Diagn. 2013;33(7):667–74. Epub 2013/04/18. pmid:23592541.
- Benn P, Cuckle H, Pergament E. Non-invasive prenatal testing for aneuploidy: current status and future prospects. Ultrasound Obstet Gynecol. 2013;42(1):15–33. Epub 2013/06/15. pmid:23765643.
- Gregg AR, Gross SJ, Best RG, Monaghan KG, Bajaj K, Skotko BG, et al. ACMG statement on noninvasive prenatal screening for fetal aneuploidy. Genet Med. 2013;15(5):395–8. Epub 2013/04/06. doi: gim201329 [pii] pmid:23558255.
- Benn P, Borell A, Chiu R, Cuckle H, Dugoff L, Faas B, et al. Position statement from the Aneuploidy Screening Committee on behalf of the Board of the International Society for Prenatal Diagnosis. Prenat Diagn. 2013;33(7):622–9. Epub 2013/04/26. pmid:23616385.
- Bianchi DW, Platt LD, Goldberg JD, Abuhamad AZ, Sehnert AJ, Rava RP, et al. Genome-wide fetal aneuploidy detection by maternal plasma DNA sequencing. Obstet Gynecol. 2012;119(5):890–901. Epub 2012/03/01. pmid:22362253.
- van den Berg C, Van Opstal D, Polak-Knook J, Galjaard RJ. (Potential) false-negative diagnoses in chorionic villi and a review of the literature. Prenat Diagn. 2006;26(5):401–8. Epub 2006/03/16. pmid:16538702.
- Kalousek DK, Dill FJ, Pantzar T, McGillivray BC, Yong SL, Wilson RD. Confined chorionic mosaicism in prenatal diagnosis. Hum Genet. 1987;77(2):163–7. Epub 1987/10/01. pmid:3653890.
- Grati FR, Malvestiti F, Ferreira JC, Bajaj K, Gaetani E, Agrati C, et al. Fetoplacental mosaicism: potential implications for false-positive and false-negative noninvasive prenatal screening results. Genet Med. 2014;16(8):620–4. Epub 2014/02/15. doi: gim20143 [pii] pmid:24525917.
- Toutain J, Van-Gils J, Horovitz J, Saura R. Comment on “QF-PCR as a substitute for karyotyping of cytotrophoblast for the analysis of chorionic villi: advantages and limitations from a cytogenetic retrospective audit of 44 727 first-trimester prenatal diagnoses”. Prenat Diagn. 2013;33(11):1115–6. Epub 2013/07/23. pmid:23868568.
- Toutain J, Epiney M, Begorre M, Dessuant H, Vandenbossche F, Horovitz J, et al. First-trimester prenatal diagnosis performed on pregnant women with fetal ultrasound abnormalities: the reliability of interphase fluorescence in situ hybridization (FISH) on mesenchymal core for the main aneuploidies. Eur J Obstet Gynecol Reprod Biol. 2010;149(2):143–6. Epub 2010/01/05. doi: S0301-2115(09)00737-4 [pii] pmid:20045588.
- Porreco RP, Garite TJ, Maurel K, Marusiak B, Obstetrix Collaborative Research N, Ehrich M, et al. Noninvasive prenatal screening for fetal trisomies 21, 18, 13 and the common sex chromosome aneuploidies from maternal blood using massively parallel genomic sequencing of DNA. Am J Obstet Gynecol. 2014;211(4):365 e1-12. Epub 2014/03/25. doi: S0002-9378(14)00270-1 [pii] pmid:24657131.
- Gil MM, Akolekar R, Quezada MS, Bregant B, Nicolaides KH. Analysis of cell-free DNA in maternal blood in screening for aneuploidies: meta-analysis. Fetal Diagn Ther. 2014;35(3):156–73. Epub 2014/02/12. doi: 000358326 [pii] pmid:24513694.
- Gerovassili A, Garner C, Nicolaides KH, Thein SL, Rees DC. Free fetal DNA in maternal circulation: a potential prognostic marker for chromosomal abnormalities? Prenat Diagn. 2007;27(2):104–10. Epub 2006/12/08. pmid:17152117.
- Vora NL, Johnson KL, Basu S, Catalano PM, Hauguel-De Mouzon S, Bianchi DW. A multifactorial relationship exists between total circulating cell-free DNA levels and maternal BMI. Prenat Diagn. 2012;32(9):912–4. Epub 2012/06/22. pmid:22717986; PubMed Central PMCID: PMC3432738.
- Rava RP, Srinivasan A, Sehnert AJ, Bianchi DW. Circulating fetal cell-free DNA fractions differ in autosomal aneuploidies and monosomy X. Clin Chem. 2014;60(1):243–50. Epub 2013/09/21. doi: clinchem.2013.207951 [pii] pmid:24046201.
- Van Opstal D, de Vries F, Govaerts L, Boter M, Lont D, van Veen S, et al. Benefits and burdens of using a SNP array in pregnancies at increased risk for the common aneuploidies. Hum Mutat. 2015;36(3):319–26. Epub 2014/12/17. pmid:25504762.
